Décrypter l'épigénome à l'aide de technologies basées sur CRISPR
La percée de CRISPR-Cas9 a accéléré notre compréhension de la génétique et ouvert la voie à la guérison de nombreuses maladies liées à l'ADN. Cet outil agit comme des ciseaux moléculaires qui peuvent être dirigés vers des sites spécifiques du génome lorsqu'ils sont guidés par une molécule analogue à un navigateur GPS : l'ARN guide. De nouvelles technologies ont adapté la machinerie CRISPR-Cas9 pour élargir son utilisation de découpeur d'ADN capable d'éditer le génome, à celui de modulateur épigénétique qui édite l'épigénome. Cette nouvelle approche permet une meilleure compréhension de l'influence de l'épigénome sur le fonctionnement cellulaire, et présente un potentiel futur en matière de traitement de maladies épigénétiques.
Adaptation de CRISPR-Cas9 pour modifier l'épigénome
Pour étudier correctement le rôle de l'épigénome dans les organismes vivants, les scientifiques ont muté le ciseau moléculaire Cas9 - de sorte qu'il ne puisse plus couper l'ADN - et l'ont rebaptisée Cas9 désactivée, ou défunte (dCas9). Les composantes essentielles de la machinerie CRISPR-dCas9 restant intactes, l'outil peut toujours reconnaître un ARN guide afin d’être dirigé vers un emplacement spécifique du génome.
Les scientifiques exploitent la fonction de l'enzyme défunte pour modifier l'épigénome en la fusionnant avec des modulateurs épigénétiques de leur choix. Essentiellement, les modificateurs d'histone ou de méthylation de l'ADN s'accrochent à la dCas9, et l'ensemble de la machinerie suit l'ARN guide conçu sur mesure pour cibler une séquence génomique spécifique. Une fois que l'ARN guide a trouvé la séquence d'ADN cible, le modulateur épigénétique va remodeler l'épigénome à cet endroit en utilisant sa fonction normale.
Modèles de souris conçus pour exprimer des éditeurs de l’épigénome
Les études sur l'édition de l'épigénome dans les organismes vivants sont principalement réalisées chez la souris. Comme les souris n'expriment pas le système d'édition de l'épigénome CRISPR-dCas9, les scientifiques doivent génétiquement intégrer sa séquence codante dans des régions qui ne perturberont pas le fonctionnement normal du génome. Un promoteur doit être placé devant la séquence codante pour permettre son expression. Ce promoteur peut être constitutif, pour exprimer la machinerie d’édition de l’épigénome dans tous les types cellulaires, ou être spécifique à un type de cellule ou de tissu. Pour cibler le remodelage épigénétique sur des segments d'ADN spécifiques, l'ARN guide peut également être intégré au génome de la souris, ou être livré aux cellules à l'aide de virus inoffensifs. Ces stratégies novatrices permettent aux scientifiques de répondre à de nombreuses questions pressantes dans le domaine de l'épigénétique in vivo.
Comment les marques épigénétiques contribuent-elles aux différents types cellulaires et tissulaires?
La plupart des types cellulaires sont composées de la même séquence d'ADN. Les modifications épigénétiques qui ornent l'ADN influencent la façon dont les cellules acquièrent les spécificités d’un type cellulaire en particulier. Les régions régulatrices du génome tels que les promoteurs peuvent être distingués par leurs modifications épigénétiques. Dans la plupart des cas, les promoteurs actifs dans des cellules spécifiques sont marqués par des niveaux élevés de H3K4me3 et/ou de H3K27ac tandis que les promoteurs inactifs sont marqués par des niveaux élevés de H3K27me3 et/ou de H3K9me3. Des études portant sur la caractérisation des marques épigénétiques à travers le génome ont permis d'identifier des signatures épigénétiques spécifiques aux différents types de cellules et de tissus. Par exemple, les promoteurs de gènes importants pour le fonctionnement des neurones porteront la marque active H3K4me3 dans le tissu cérébral mais pas dans le tissu pulmonaire. Néanmoins, ces observations restent majoritairement associatives. On ne sait pas nécessairement si ces signatures épigénétiques régulent l’activité des gènes de manière causale.
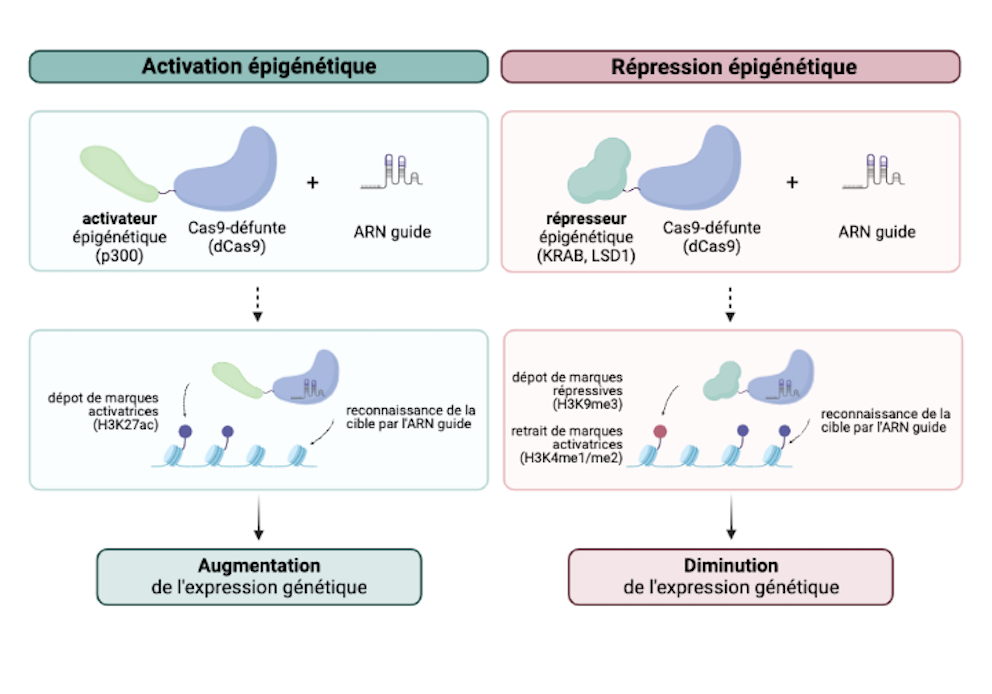
Grâce à l’utilisation de systèmes d’édition de l'épigénome dans des modèles de souris, les scientifiques peuvent déterminer comment les modifications épigénétiques au niveau des promoteurs de tissus spécifiques influencent directement l'expression des gènes. Dans une étude, Gemberling et Siklenka et al. ont attaché à dCas9 soit le répresseur transcriptionnel KRAB, qui favorise le dépôt de marques répressives (méthylation de l'ADN et H3K9me3), soit l’activateur transcriptionnel p300, qui dépose la marque active H3K27ac. Ils ont génétiquement intégré les éditeurs de l’épigénome dans le génome de la souris de façon à ce qu'ils ne soient exprimés que dans le foie.
L'équipe a ciblé la machinerie activatrice dCas9-p300 sur Pdx1, un gène qui peut entraîner un diabète précoce lorsqu'il est défectueux. Ils ont constaté que l'outil dCas9-p300 était capable de déposer la marque activatrice H3K27ac spécifiquement sur le promoteur de Pdx1 et ainsi causer une augmentation directe de l'expression du gène. Toujours dans le foie, ils ont dirigé la machinerie répressive dCas9-KRAB vers Pcsk9, un gène impliqué dans la réduction du cholestérol. Là encore, leur ciblage était très spécifique; Ils ont observé une diminution de l'expression de Pcsk9 causée par l’augmentation ciblée de la marque répressive H3K9me3 au niveau de son promoteur. Ces résultats indiquent que dCas9-p300 et dCas9-KRAB peuvent activer et désactiver des gènes de façon robuste et tissu-spécifique en ciblant les promoteurs.
Comment décoder le génome régulateur grâce aux systèmes dCas9?
Les amplificateurs (enhancers en anglais) sont des séquences d'ADN qui régulent avec précision le niveau de transcription d'un gène situé à proximité ou à une distance éloignée dans le génome. En général, plusieurs amplificateurs régulent l'expression d'un seul gène, souvent de manière tissu-spécifique. De plus, ils ont généralement des effets redondants, donc l’inhibition d'un seul amplificateur pourrait ne pas causer d’effet sur l’expression du gène qu’il régule. Il est intéressant de noter que, comme les promoteurs, les amplificateurs peuvent être distingués par leurs modifications épigénétiques. Les amplificateurs actifs présentent généralement des niveaux élevés de H3K4me1 et de H3K27ac, mais de faibles niveaux de H3K4me3.
De nombreux groupes ont profité de ces outils d'édition de l'épigénome pour modifier les marques épigénétiques sur des amplificateurs spécifiques et pour ensuite observer comment le gène régulé par l'amplificateur répond à ces changements. Gemberling et Siklenka et al. ont réussi à activer l’amplificateur cerveau-spécifique flanquant le gène Fos en utilisant leur modèle de souris dCas9-p300. L’augmentation de H3K27ac sur cet amplificateur a engendré une augmentation de l'expression du gène Fos uniquement dans le cerveau.
Dans une autre étude, Li et al. ont exprimé chez la souris deux systèmes dCas9 répresseurs simultanément, soit dCas9-KRAB et un dCas9-LSD1, pour interroger la fonction des amplificateurs. LSD1 est une déméthylase d’histone qui supprime les marques H3K4me1 et H3K4me2 et est connue pour sa capacité à inhiber les amplificateurs. Li et al. ont étudié les amplificateurs qui régulent les gènes impliqués dans la formation de la moelle osseuse (myélopoïèse) en se concentrant sur Cebpa, un gène impliqué dans la leucémie. Avec leur système dCas9 répresseur double, ils ont ciblé 4 amplificateurs flanquant le gène Cebpa. Ils ont constaté que 2 des amplificateurs sont requis pour la régulation de Cebpa, dont l'un à un moindre degré que l'autre, tandis que les 2 autres n’ont aucun effet sur Cebpa. Ces études mettent en évidence la complexité du génome régulateur et laissent entrevoir comment les outils d'édition de l'épigénome peuvent être utilisés pour en faciliter le décryptage.
La pointe de l'iceberg
Les nouvelles avancées dans les technologies d'édition de l'épigénome ont permis aux scientifiques de se munir d'outils leur permettant de mieux analyser les mécanismes épigénétiques. Avec des centaines de types cellulaires et de modifications épigénétiques, ainsi que des milliers de loci régulateurs, acquérir une compréhension plus approfondie de notre épigénome de façon tissu- et cellule-spécifique est une tâche ardue. Néanmoins, une meilleure caractérisation de la régulation épigénétique grâce au développement d'outils d'édition de l'épigénome constitue un premier pas vers l’utilisation de ces technologies dans un contexte thérapeutique.
Apprendre Encore Plus
- Technologies CRISPR pour une édition précise de l'épigénome: https://pubmed.ncbi.nlm.nih.gov/33420494/
- CRISPR 101 : Épigénétique et édition de l'épigénome: https://blog.addgene.org/crispr-101-editing-the-epigenome
- Modificateurs et modulateurs de la chromatine : régulateurs de la différenciation cellulaire: https://pubmed.ncbi.nlm.nih.gov/24366184/
Nous tenons à remercier Dr. Serge McGraw de l’Université de Montréal pour son avis d'expert sur cet article et Elizabeth Elder pour la traduction Française.
Image créée danswww.BioRender.com