Benjamin Lebeau a obtenu son baccalauréat du département d'anatomie et de biologie cellulaire de l'Université McGill. Il étudie maintenant comme étudiant au doctorat dans le laboratoire du Dr Michael Witcher à l'Institut Lady Davis de l'Université McGill. Ses recherches portent sur l'impact oncogénique et épigénétique des mutations ou de la perte de CTCF dans le cancer du sein. Ses projets étudient actuellement les changements de conformation de la chromatine à l'origine de la progression du cancer du sein suite à la perte d'hétérozygotie de CTCF et un nouvel outil bioinformatique pour étudier la modification des motifs d'ADN suite à la mutation de CTCF. Ensuite, il prévoit de déménager à Singapour pour poursuivre des études postdoctorales qui font le pont entre l'intelligence artificielle et l'épigénétique. En savoir plus sur Benjamin.
Sur quoi travailles-tu ?
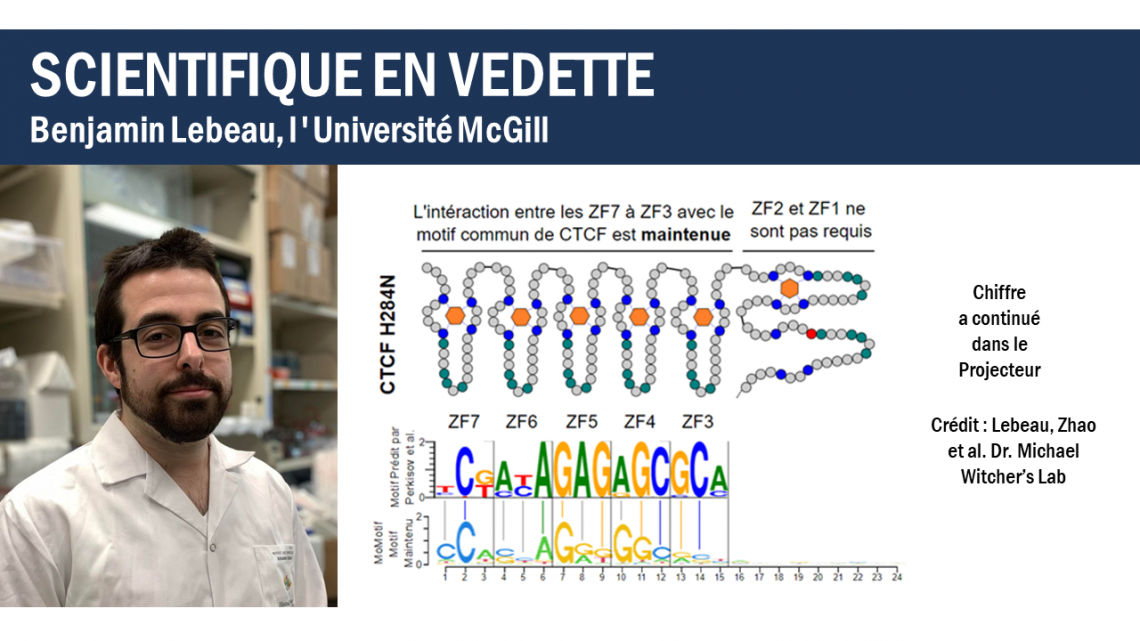
La définition de l'impact des mutations sur la reconnaissance des motifs d'ADN dépend fortement des outils bio-informatiques qui définissent les éléments se liant à l'ADN. Cependant, les outils classiques d'analyse de motifs restent limités dans leur capacité à identifier des changements subtils aux motifs de liaison complexes entre des conditions distinctes. Pour surmonter cette limitation, nous avons développé un nouvel outil, MoMotif, qui facilite une identification sensible, avec une résolution à la paire de bases près, des transformations complexes ou subtiles des motifs d’ADN, discernées à partir des résultats de ChIP-seq. Nous avons utilisé MoMotif pour définir le motif de reconnaissance non caractérisé du doigt de zinc 1 (ZF1) de CTCF et pour définir plus précisément l'impact de la mutation de CTCF ZF1 sur son association avec la chromatine. Les mutations de CTCF ZF1 sont exclusives au cancer du sein et sont associées à des métastases et à une résistance thérapeutique, mais les mécanismes sous-jacents ne sont pas clairs. À l'aide de MoMotif, nous avons identifié une extension du motif d’ADN reconnu par CTCF, nécessitant un ZF1 fonctionnel pour s’y lier de manière appropriée. En utilisant une combinaison de ChIP-Seq et d'ARN-Seq, nous découvrons que l'incapacité à se lier à ce motif étendu entraîne un programme transcriptionnel altéré associé aux phénotypes oncogènes observés cliniquement. Notre étude démontre que MoMotif est un nouvel outil pour l'analyse comparative de ChIP-seq et la caractérisation des contacts ADN-protéine.

Crédit: Lebeau, Zhao et al. Dr. Michael Witcher’s Lab
D'où viens-tu? Qu'est-ce qui te manque de ta ville/pays d’origine ?
Je viens de Joliette dans la province de Québec. Étant donné que c’est à moins de 2h de route d’où je vis présentement, j’y retourne souvent, donc je n’ai pas le temps de m’ennuyer!
Dans quelle ville vis-tu actuellement et qu'est-ce que tu aimes le plus dans cette ville ?
J’habite à Montréal. C’est une ville très vivante et décontractée avec beaucoup d’espaces verts, de bars et de restaurants. Donc il y a toujours quelque chose de plaisant à faire à Montréal!
De quelles manières te déconnectes-tu du travail, de la science, du milieu académique ? As-tu un exemple préféré ?
J’aime courir des longues distances et être actif. C’est plaisant de pratiquer une discipline physiquement demandante (contrairement à la science, qui travaille plus le cerveau) pour laquelle je peux m’investir et chercher constamment à m’améliorer. Aussi, ça m’énergise et me relax mentalement, tout en utilisant efficacement les calories accumulés dans les bars et restos mentionnés plus haut.
As-tu des talents particuliers en dehors de la recherche dont peu de personnes dans ton entourage académique sont au courant ?
Je n’appellerais pas ça un talent, mais j’aime bien en apprendre plus sur les techniques de cuisine et la science derrière les saveurs et les textures. Donc, au fil du temps, je suis devenu un cuisinier assez polyvalent.
As-tu des recommandations pour un livre, une série TV, un film ou un documentaire ?
Je dirais la série de manga de Eiichiro Oda qui est publié hebdomadairement depuis environ 20 ans : One Piece. Il y a déjà plus de 1000 chapitres, soit des centaines d’heures de lecture, mais les personnages et l’histoire en valent la peine!
Qu'est-ce qui t’a poussé à devenir chercheur ?
J’aime constamment apprendre, les défis intellectuels et résoudre des problèmes complexes. Donc, mon intérêt pour la recherche et la science m’est venu assez naturellement.
Si tu n’étais pas chercheur, que penses-tu que tu serais devenu ou qu’est-ce tu aurais aimé être ?
Je travaillerais probablement en économie et finance internationales.
Qu'est-ce qui a suscité ton intérêt pour l'épigénétique ?
La régulation épigénétique de l’expression des gènes est très dynamique et implique des réseaux incroyablement complexes d’effecteurs. Il y a encore tellement d’aspects à découvrir et approfondir! En d’autres termes (hautement scientifiques), je trouve l’épigénétique très cool.
Si tu pouvais donner un conseil à un « doctorant de première année », quel serait-il ?
Étant donné que j’ai commencé en 2018, je dirais : « Va au plus de conférences en personne possible, dans 2 ans elles seront très rares!
Comment aimerais-tu voir ton domaine de recherche évoluer dans l'avenir ?
J'aimerais voir plus de collaborations interdisciplinaires. Des travaux récents ont montré comment la recherche en biophysique, en imagerie cellulaire ou en intelligence artificielle/modélisation informatique, pour n'en citer que quelques-unes, peut être appliquée à l'étude de l'épigénétique. L'intégration de ces techniques ensemble pourrait nous éclairer sur de nouvelles approches pour cibler thérapeutiquement ces processus de régulation avec une plus grande prédictivité entre les modèles et les organismes.