Oncohistones : Conducteurs épigénétiques du cancer et au-delà
par Wajih Jawhar
Les mutations génétiques ayant un impact sur les processus épigénétiques sont fréquentes et peuvent être pathognomoniques dans le cancer. Une catégorie de ces mutations modifie directement les histones, donnant naissance à des « oncohistones ». Ces oncohistones favorisent la transformation cellulaire en altérant le profil des modifications post-traductionnelles (PTM) des histones à l'échelle du génome. L'analyse fonctionnelle des oncohistones a permis de mieux cerner les mécanismes pathologiques et d'approfondir notre compréhension du contrôle épigénétique des processus cellulaires.
H3K27M:
L'une des oncohistones les plus étudiées est l'H3K27M, où le 27e résidu d'acide aminé de la queue de l'histone H3 est modifié de la lysine (K) à la méthionine (M) (Fig. 1). Cette mutation peut se produire dans n'importe quelle variante de l'histone H3, qu'elle soit canonique (H3.1/H3.2) ou non canonique (H3.3). L'H3K27M remodèle le profil chromatinien en inhibant l'activité du complexe répressif Polycomb 2 (PRC2), un complexe protéique clé responsable du dépôt des marques mono-, di- et tri-méthyl sur l'H3K27. En ajoutant ces marques répressives, PRC2 orchestre le développement normal dans l'espace et dans le temps en régulant l'expression de gènes clés du développement au sein des cellules souches, contribuant ainsi à la formation des quelque 300 types de cellules différenciées qui composent le corps humain (Harutyunyan et al., 2019 ; Yu et al., 2019).
Les oncohistones H3K27M ont été initialement découvertes et caractérisées dans des tumeurs cérébrales pédiatriques agressives de la ligne médiane, collectivement appelées « gliomes diffus de la ligne médiane, avec altération K27 ». L'intérêt croissant et l'étude approfondie de ces oncohistones ont conduit à leur identification dans d'autres cancers du système nerveux central (SNC), tels que les épendymomes PFA, et, plus rarement, dans des sous-ensembles de cancers des globules blancs (leucémie myéloïde aiguë) et des cancers des os (ostéosarcome) (Krug et al., 2021 ; Salomoni et al., 2023).

Tout au long du développement, le complexe PRC2 est initialement recruté avec l'aide de protéines accessoires (par exemple, MTF2 et JARID2) pour déposer H3K27me3 dans des régions riches en CpG appelées îlots CpG. Ce processus, désigné sous le terme de « nucléation », représente l'établissement initial de foyers de H3K27me3 sur la chromatine. PRC2 est ensuite activé de manière allostérique en liant son produit H3K27me3, ce qui induit un « étalement » rapide de H3K27me2 et H3K27me3 sur la chromatine adjacente et de manière distale via des interactions à longue portée avec les hubs de nucléation (Oksuz et al., 2018).
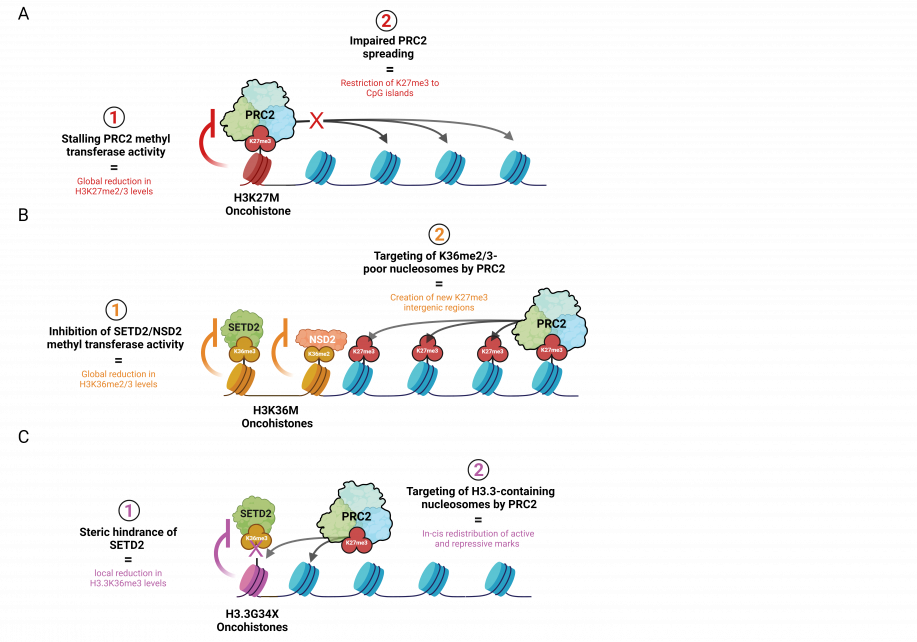
Cependant, dans les cellules qui acquièrent H3K27M, le blocage de PRC2 entraîne une diminution globale de la di- et de la tri-méthylation de H3K27, ainsi qu'un étalement à partir des îlots CpG. Étant donné que la catalyse de H3K27me3 est enzymatiquement plus exigeante que celle de H3K27me2, ces effets sont plus prononcés pour la première marque d'histone (Fig. 2A). Sur le plan fonctionnel, ce phénomène crée un profil épigénomique qui favorise un état prolifératif similaire à celui des cellules souches, associé à la transformation (Harutyunyan et al., 2019 ; Boileau et al., 2019).
H3K36M
Similairement à la lysine 27, le résidu voisin de la lysine 36 (K36) est également un point chaud pour les mutations K-to-M dans certains sous-types de cancer. La plupart de nos connaissances sur les oncohistones K36M proviennent de cancers mésenchymateux tels que le chondroblastome, le chondrosarcome et le sarcome pléomorphe indifférencié, qui hébergent des mutations H3.1/H3.3K36M (Lu et al., 2016 ; Behjati et al., 2013). Bien que la mutation K36M ait été initialement découverte dans un sous-ensemble de carcinomes épidermoïdes de la tête et du cou (Schapira et al., 2014), des travaux plus récents ont démontré son rôle dans l'évasion immunitaire associée à ces types de cancer (Li et al., 2022).
Le H3K36M agit comme un puissant inhibiteur des protéines NSD2 et SETD2, responsables du dépôt des marques d'histone H3K36me2 et H3K36me3, respectivement. La première PTM présente une large distribution à travers le génome et délimite les régions intergéniques, tandis que la seconde est enrichie dans les corps des gènes activement transcrits. En plus de favoriser l'expression des gènes, la méthylation de K36 s'oppose au silençage médié par PRC2 en perturbant l'interaction entre la queue de l'histone H3 et le site actif d'EZH2, la sous-unité catalytique de PRC2 (Lu et al., 2016 ; Fang et al., 2016 ; Muller et al., 2020). Étant donné l'antagonisme fonctionnel entre les méthylations K36 et K27, la réduction de H3K36me2/3 à l'échelle du génome crée un nouvel environnement d'histones plus permissif pour le ciblage de PRC2.
Dans une étude examinant cette relation inverse dans le contexte des sarcomes K36M, l'expression de K36M n'a pas entraîné d'augmentation des niveaux géniques de H3K27me3, mais a plutôt créé de nouveaux domaines intergéniques H3K27me3 dans les régions ayant perdu H3K36me2 (Fig. 2B) (Lu et al., 2016). Par conséquent, la redistribution ou la « dilution » de H3K27me3 génique a conduit à une dilution correspondante du complexe répressif canonique de polycomb 1 (cPRC1), qui agit comme lecteur de H3K27me3. Lors du recrutement de cPRC1 sur les cibles de polycomb délimitées par H3K27me3, cPRC1 monoubiquitine la lysine 119 de l'histone H2A (H2A119ub), favorisant ainsi la formation de l'hétérochromatine et la répression des gènes (Gao et al., 2013). Dans les progéniteurs mésenchymateux exprimant H3K36M, la dilution de cPRC1 de ces cibles entraîne une dé-répression aberrante de gènes qui entravent la différenciation des cellules souches et favorisent la sarcomagenèse (Lu et al., 2016 ; Fang et al., 2016).
H3G34X
Une caractéristique commune des mutations K36M et K27M est leur présence sur des résidus lysine qui subissent des modifications post-traductionnelles (PTM). Cependant, tous les 59 résidus d'acides aminés de la queue de l'histone H3 ne subissent pas ces modifications. Il est intéressant de noter que des mutations sur des résidus chimiquement non modifiés, tels que la glycine 34 (G34), sont également impliquées dans des maladies comme le cancer.
La glycine 34, située à deux résidus de la lysine 36, peut abriter quatre mutations oncohistones différentes : H3 G34W, G34L, G34R et G34V, qui transforment respectivement la glycine en tryptophane, leucine, arginine ou valine (Fig. 1). Les deux premiers changements d'acides aminés (G34W/L) se produisent le plus souvent dans les tumeurs des tissus conjonctifs (mésenchymateux), tandis que les deux derniers (G34R/V) se rencontrent généralement dans les tumeurs du cortex cérébral, connues sous le nom de « gliomes hémisphériques diffus, altérés par G34 » (Behjati et al., 2013 ; Khazaei et al., 2020).
Contrairement aux mutations H3K27M et H3K36M, qui modifient chimiquement le comportement des enzymes par inhibition ou modulation allostérique, les oncohistones G34X fonctionnent par interférence physique. En effet, la glycine, un acide aminé de petite taille, est remplacée par un résidu volumineux tel que le tryptophane, créant ainsi un obstacle stérique. De plus, les mutations G34X se produisent exclusivement sur les histones H3.3, ce qui pourrait indiquer que leur emplacement sur la chromatine est crucial pour la pathogenèse. Lorsque l'enzyme SETD2 est recrutée pour tri-méthyler K36, le résidu désormais volumineux bloque physiquement cette réaction. Ce phénomène est connu sous le nom d'« inhibition stérique », se référant à l'arrangement spatial d'une molécule. Cela entraîne une perte locale de tri-méthylation K36 au niveau des gènes activement transcrits, provoquant une méthylation aberrante de K27 par PRC2 et, par conséquent, une réduction du silence de ces loci (Fig. 2C).
Dans les tumeurs osseuses à cellules géantes (GCTB), la mutation H3.3G34W induit des changements transcriptionnels qui entraînent une résorption osseuse anormale (dégradation osseuse) et une invasion locale des tissus osseux environnants, caractéristiques de cette entité tumorale (Jain et al., 2020 ; Khazaei et al., 2020). D'autres études sur les oncohistones G34 continuent d'élucider leur fonction dans diverses maladies, telles que la neurodégénérescence, et leurs mécanismes pathologiques établissent un lien entre la méthylation aberrante des histones et les schémas de méthylation de l'ADN (Khazaei et al., 2023).
Dans l'ensemble, cet équilibre complexe entre les modifications des histones, connu collectivement sous le nom de « code des histones » (Allis et Strahl, 2000), demeure un domaine de recherche actif. L'expression génique n'est pas uniquement contrôlée par la présence ou l'absence d'une marque d'histone spécifique, mais également par sa concentration et sa dilution relatives sur la chromatine, ainsi que par son interaction avec le reste de la boîte à outils épigénétique.